Plateforme genEPII : Plateforme de GENomique à visée diagnostique et EPIdémiologique des maladies Infectieuses
Plateforme de séquençage haut-débit spécialisée en microbiologie
La plateforme GenEPII (GENomique à visée diagnostique et EPIdémiologie des maladies Infectieuses) est l’une des 4 plateformes nationales de séquençage microbiologique à finalité de Santé Publique créées en 2021 par l’Agence Nationale de Recherche en Santé – Maladies Emergentes et Santé Publique France (ARNS-MIE) et par les HCL dans le cadre de la surveillance de l’évolution génétique du Sars-Cov-2.
Elle est intégrée au Pôle d’Activité Médicale Biologie et Anatomique Pathologique des HCL qui est organisé en laboratoire unique appelé Laboratoire Biologie Médicale Multi-sites (LBMMS) et positionnée au sein de l’Institut des Agents Infectieux (IAI) du GHN.
Cette plateforme réalise maintenant l’ensemble des séquençages à visée microbiologique du Centre de Biologie Nord, que ce soit dans le cadre du diagnostic ou du suivi épidémiologique.
Au niveau national, la plateforme genEPII est également au service des différents Centres Nationaux de Référence du Centre de Biologie Nord et est devenu Laboratoire de biologie médicale de référence (LBMR) pour différents paramètres.
Activités
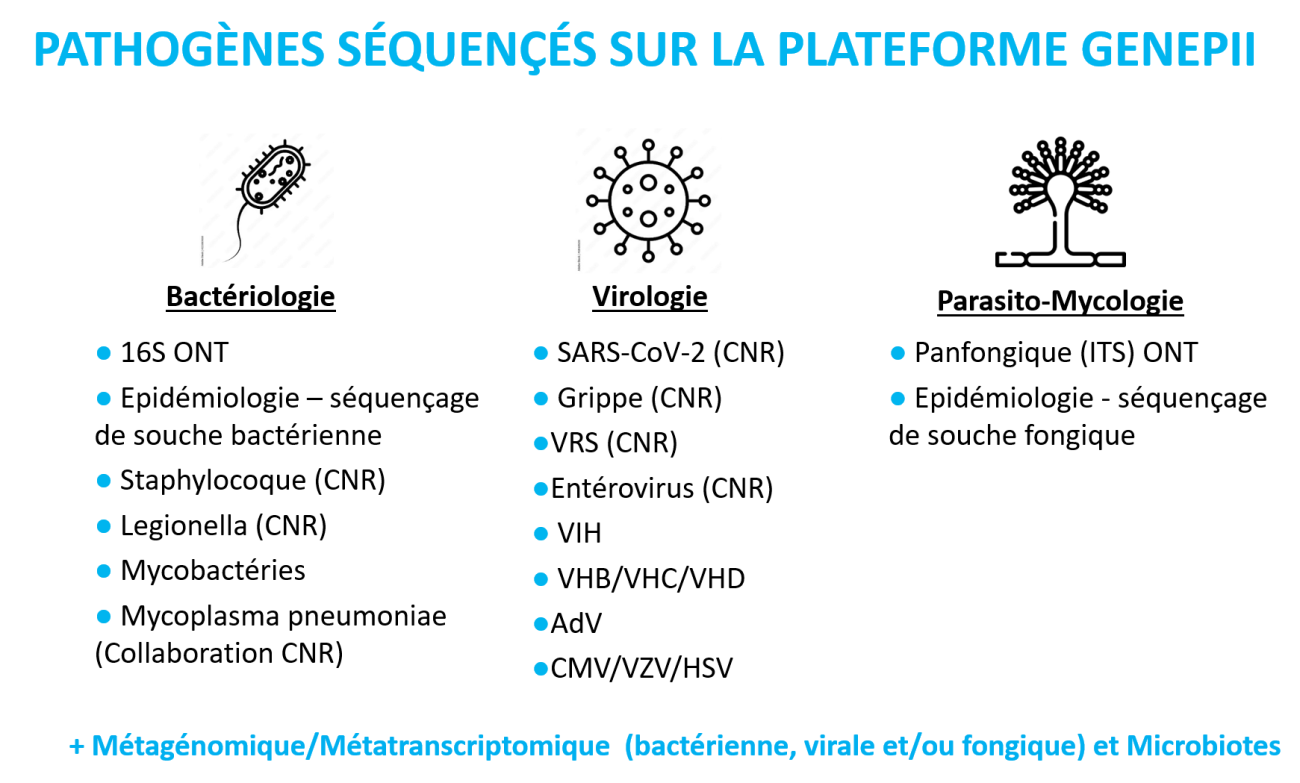
Le séquençage en microbiologie inclut le séquençage ciblé de gènes pour l’identification d’espèces au sein d’un prélèvement (gène codant l’ARNr16S bactérien, région ITS2 fongique ), de résistances aux antiviraux (ex: VIH, VHB)) ou aux antibiotiques en passant par le séquençage du génome complet du pathogène pour la surveillance épidémiologique (ex : détection d’un variant/lignage, analyse de clusters…) ou la comparaison de génomes de bactéries ou d’agents fongiques (ex : suspicion de transmission croisée), jusqu’au séquençage de l’ensemble des génomes des agents microbiens d’un prélèvement (ex : caractérisatoin du microbiote, identification de nouveau pathogène émergent après échec des méthodes de première intention).
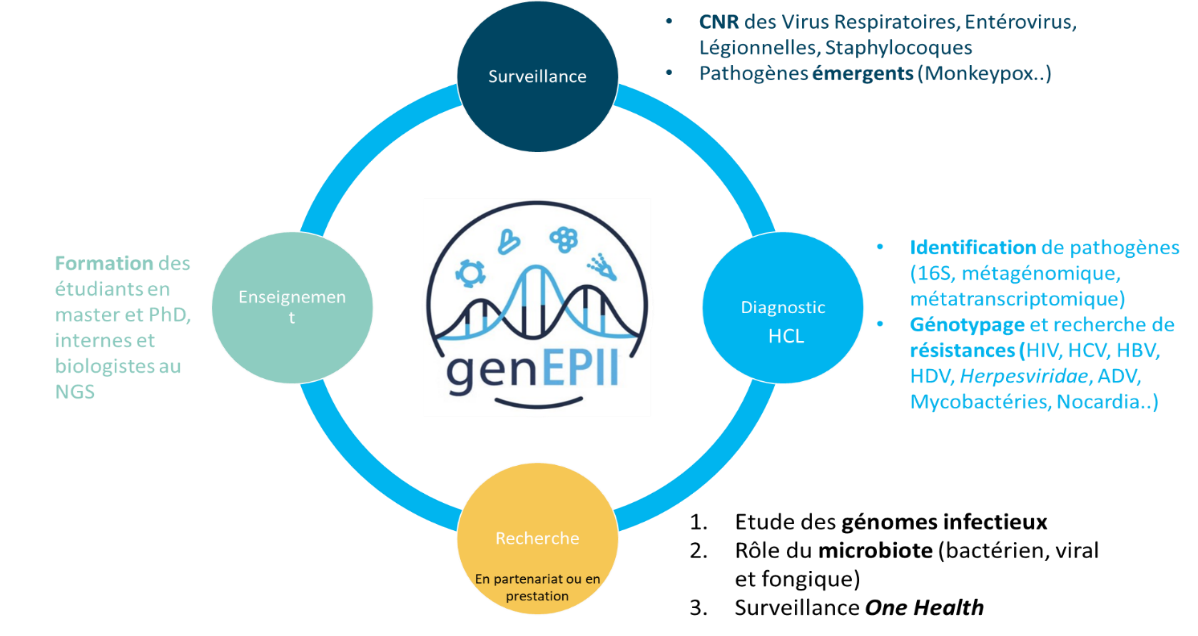
La plateforme de séquençage genEPII s’articule autour des 4 activités suivantes :
Diagnostic
- Séquençage de virus ou de bactéries pour la détection de mutation de résistance (VIH, VHC, VHB, Grippe, SARS-CoV-2, CMV, HSV, Mycobacterium tuberculosis, Mycoplasma pneumoniae, Legionella, Staphylococcus aureus)
- Détection et identification bactérienne par PCR 16S/universelle puis séquençage NGS
- Détection et identification bactérienne par PCR ITS/panfongique puis séquençage NGS
- Diagnostic de recours et identification de pathogènes émergents (techniques sans a priori recherchant l’ensemble des pathogènes (viraux, bactériens, fongiques et parasitaires) ; GenEPII est devenu LBMR (Laboratoire de biologie médicale de référence) pour cette activité
- Métagénomique shotgun (recherche et identification de l’ensemble des ADN présents dans un échantillon)
- Métatranscriptomique (recherche et identification de l’ensemble des ARN présents dans un échantillon)
Surveillance épidémiologique
- L’activité de surveillance épidémiologique des 4 Centres Nationaux de Référence (CNR des staphylocoques, des légionelles, des virus respiratoires et des entérovirus/parechovirus), ainsi que du Centre régional des Mycobactéries (LBMR de la tuberculose et des Nocardia) et le laboratoire de mycologie. Cette activité de surveillance concerne depuis fin 2023 le séquençage du VRS pour le suivi d’éventuelles résistances au Beyfortus.
- L’investigation des épidémies et cas groupés nosocomiaux au sein des HCL par séquençage des souches bactériennes et fongiques.
- GenEPII est par ailleurs devenu LBMR de Génomique comparative pour l’étude de la diffusion et de la transmission des pathogènes hors CNR sur isolats et échantillons d’origine clinique, environnementale et animale
Formation
- Accueil des stagiaires de Masters, des externes en médecine et des internes en biologie médicale
- Formation continue des biologistes
Recherche
- Le microbiote par métataxonomique (séquençage ciblé de l’ADN 16S et ITS pour l’analyse du microbiote bactérien et fongique) pour des projets de recherche translationnelle en lien avec le GEM (Groupe d’Etude du Microbiote) des HCL.
- Le développement de la métagénomique et métatranscriptomique pour l’étude du microbiote dans son ensemble et de la réponse de l’hôte
- La veille technologique constante sur le NGS (R&D).
- L’évolution intra-hôte du SARS-CoV-2 dans diverses populations et suite aux traitements ou vaccins.
- Lien avec les projets PHAG-ONE et PHAGEinLYON pour une meilleure caractérisation moléculaire, l’identification, et l’analyse bioinformatique des bactériophages composant les banques de bactériophages potentiellement utiles en thérapeutiques
Equipements
Pré-traitement - Extraction des acides nucléiques
- 1 Prescellys (Bertin Technologies)
- 2 Starlett (Hamilton)
- 2 MGI (BGI)
Préparation des amplicons en amont du séquençage (Pré-PCR)
- 1 Mosquito HV (SPTLabtech)
- 1 Dragonfly Discovery (SPTLabtech)
Préparation des banques de séquençage (Post-PCR)
- 2 Mosquito HV (SPTLabtech) pour la préparation des banques de type DNAPrep (Illumina)
- 1 Dragonfly Discovery (SPTLabtech)
- 1 DreamPrep (Tecan) dédié à la métatranscriptomique
- 1 EpMotion (Eppendorf) pour la préparation des banques de séquençage ONT et la capture.
Séquenceurs
- 1 NextSeq550 (Illumina)
- 1 NovaSeq 6000 (Illumina)
- 1 Gridion (ONT) et plusieurs minION (ONT)
Informatique
- 4 serveurs de calcul en miroir: CPU Intel Xeon Gold 6242R 20C/40T, 768GB RAM chacun, 20T de stockage chaud.
- 1 serveur de calcul, CPU Intel Xeon Gold 5317 24C/48T, 2xGPU Ampere A30, 192GB RAM, 10T de stockage chaud.
- 1 serveur possédant des cartes GPU
- 500 To d’espace de stockage long terme




Perspectives d’évolution de la plateforme
- La surveillance du SARS-CoV-2 s’est stabilisée depuis 2023 et la plateforme est passée à la deuxième étape du projet EMERGEN qui est d’assurer la surveillance moléculaire des pathologies infectieuses émergentes (virales, bactériennes, fongiques, parasitaires) avec le déploiement du séquençage pour d’autres pathologies (ex : arbovirus, Mycoplasma pneumoniae).
- Les évolutions prévues sont motivées par la volonté de proposer selon les questions posées (analyse des plasmides, circularisation du génome, facilité de l’assemblage de novo, microbiote) un séquençage long-reads de précision notamment pour les études de résistome et de virulome, protéomique à révélation NGS, validation de biomarqueurs par quantification absolue de pathogènes par la technologie ddPCR.
- Perspectives pour la recherche translationnelle nécessitant du NGS pour les HCL, et s’ouvrir à des collaborations académiques et industrielles à l’échelle locale et nationale.
- GenEPII fait partie de PETRA (Plateforme d’Expertise technologique TRAnslationnelle et transdisciplinaire) avec une volonté d’ouverture de cette activité d’expertise sur le NGS aux équipes cliniques et de recherche française.
- Liens avec Shape-Med@Lyon pour des projets transdisciplinaires faisant intervenir le NGS.
- Laboratoire de biologie médicale et d’anatomie pathologique - Page
- Institut des agents infectieux - Rubrique
- Centre National de Référence virus des infections respiratoires (dont la grippe et le SARS-CoV-2) - Rubrique
- Centre National de Référence des légionelles - Rubrique
- Centre national de référence des staphylocoques - Rubrique
- Séquençage des variants du SARS-CoV-2 : les HCL en première ligne - Actualité
- AURAGEN, plateforme de séquençage du génome à très haut débit - Page
- Métatranscriptomique, la nouvelle frontière du diagnostic - Actualité
- Le séquençage génomique, profileur de virus - Actualité
Responsables
Pr Sophie Jarraud (Bactériologie), Dr Laurence Josset (Virologie), Dr Jean Menotti (Myco-parasitologie)
Contact
Centre de Biologie Nord – Institut des Agents Infectieux
Bâtiment T - 103, Grande-Rue de la Croix-Rousse, 69317 LYON Cedex 04
Tél. standard IAI : + 33 (0)4 72 07 11 11
Fax : +33 (0)4 72 07 18 42
Adresse mail générique sécurisée : ms-lbmms-cbn-iai@chu-lyon.aura.mssante.fr [à utiliser avec une adresse mail sécurisée de santé]
- Laboratoire de biologie médicale et d’anatomie pathologique - Page
- Institut des agents infectieux - Rubrique
- Centre National de Référence virus des infections respiratoires (dont la grippe et le SARS-CoV-2) - Rubrique
- Centre National de Référence des légionelles - Rubrique
- Centre national de référence des staphylocoques - Rubrique
- Séquençage des variants du SARS-CoV-2 : les HCL en première ligne - Actualité
- AURAGEN, plateforme de séquençage du génome à très haut débit - Page
- Métatranscriptomique, la nouvelle frontière du diagnostic - Actualité
- Le séquençage génomique, profileur de virus - Actualité
